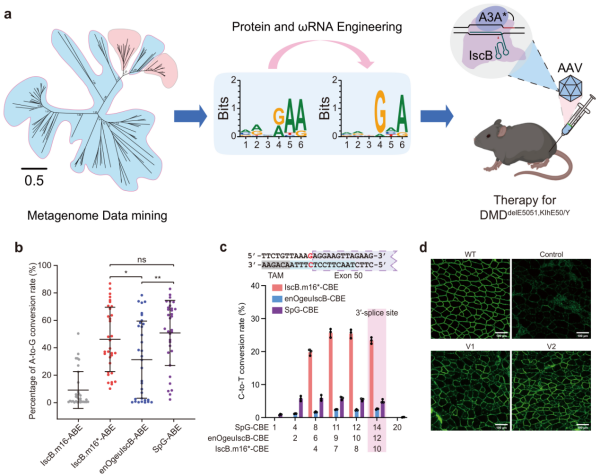
8月15日,中国科学院脑科学与智能技术卓越创新中心杨辉团队与复旦大学附属眼耳鼻喉科医院黄锦海团队等合作,在《自然-化学生物学》(Nature Chemical Biology)上发表了题为Engineered IscB–ωRNA system with expanded target range for base editing的研究论文。该研究通过宏基因组数据挖掘,鉴定出19种具有不同TAM范围的新型IscB-ωRNA系统;综合利用RNA结构优化、蛋白质工程化改造、流式细胞术、脱靶检测等技术手段,获得了在人类细胞内具有靶标识别范围广、更高效编辑活性的IscB系统(IscB.m16*);通过融合脱氨酶结构域,进一步开发出基于新型IscB的迷你型腺嘌呤和胞嘧啶碱基编辑器,并在哺乳动物细胞和小鼠疾病模型中包括SpCas9-BE无活性的疾病位点上验证了其碱基编辑效率和靶标识别能力,为未来精准基因治疗临床应用提供了支持。
CRISPR/Cas9系统自发现以来,得到了快速发展,并被广泛应用于生命科学基础研究、基因治疗、动植物育种改良等领域。基于Cas9切口酶(nCas9)与脱氨酶结构域/糖基化酶(MPG或UNG)的融合成的碱基编辑器(ABE、CBE、gGBE、gTBE),可高效实现A-to-G、C-to-T、C-to-G、G-to-C/T及T-to-G/C的碱基替换,为纠正突变的疾病位点提供了精准高效的基因编辑工具。然而,由于Cas9的体积过大,基于nCas9的碱基编辑器难以实现单个腺相关病毒(AAV)的包装递送,限制了在体基因编辑的发展应用。近年来,一系列紧凑型的Cas9蛋白、Cas12f系列同源物及其祖先蛋白TnpB被报道,但由于编辑活性有限或者缺乏HNH结构域而难以改造为缺口酶,限制了其用于碱基编辑器的开发。
IscB-ωRNA系统需要严格的6位碱基靶序列邻近基序(TAM)来识别目标DNA,且识别位点有限。因此,开发靶标识别范围更广的高效小型IscB碱基编辑器较为重要。
该研究在200GB的宏基因组数据库中挖掘出19个未被表征的新型IscB系统,采用细菌耗竭实验鉴定相应的TAM序列。进一步,研究利用荧光报告系统,筛选出10个具有真核细胞活性的IscB系统。其中,IscB.m16表现出最高的编辑活性。为提高IscB.m16系统的活性并拓宽TAM范围,研究对IscB.m16核酸酶进行RuvC结构域的精氨酸扫描突变和TAM识别相关位点的饱和突变,并对其ωRNA进行茎环截短和碱基替换的优化改造。通过多轮迭代的高通量荧光报告系统筛选,研究获得了编辑活性高和TAM范围宽的IscB.m16变体。细菌消耗TAM实验发现,相较于野生型IscB.m16,TAM从MRNRAA扩展到NNNGNA。
在此基础上,该研究构建了迷你型腺嘌呤碱基编辑器和胞嘧啶碱基编辑器。哺乳动物细胞中,迷你型腺嘌呤碱基编辑器的碱基编辑效率与SpG-ABE效率相当,高于已报道的enOgeuIscB-ABE,具有更广的TAM兼容性。在人源化杜氏肌营养不良症(DMD)小鼠疾病模型中,单AAV包装的胞嘧啶碱基编辑器经注射至肌肉组织后,将小鼠肌纤维中抗肌营养不良蛋白水平恢复至野生型小鼠的40%,为DMD患者提供了有希望的基因治疗策略。
该研究通过挖掘和优化改造新型IscB系统,开发出靶向范围更广、活性更高、特异性更好的迷你型碱基编辑工具,在基于AAV的基因治疗应用中展现出潜力。
 客服热线:
客服热线: