波士顿大学、维也纳大学和奥尔堡大学的联合研究团队研发了一种受激拉曼散射双光子荧光原位杂交技术(SRS-FISH),该技术能够在研究医学和环境样本中微生物组的成像平台上以单细胞的尺度进行高通量代谢和身份识别。SRS-FISH 的成像速度非常快,每个细胞仅需10 到 100 毫秒。
研究人员使用 SRS-FISH检测了 30,000 多个单个细胞对人体肠道微生物组中各种黏膜糖的代谢反应。他们利用源于同位素检测的拉曼波段的受激拉曼散射 (SRS) 信号,可最大限度地提高细胞中的同位素标记含量,以此检测直径约为 1μm 的细胞内的低浓度代谢物。该系统能够使用单个激光光源通过双光子 FISH 实现 SRS 代谢成像。以上的改进有效促进了集成高通量平台SRS-FISH的发展,它结合了 SRS 用于单细胞稳定同位素探测以及双光子 FISH 的优势,能够快速识别细胞并且具有很高的灵敏度。
由于SRS-FISH成像速度非常快,能够将复杂微生物群落中的代谢和身份识别联系起来,有效弥补了现有技术的空白。

图1 SRS-FISH结合了拉曼散射显微镜的优势,用于单细胞稳定同位素探测和双光子荧光原位杂交,可快速精确地识别细胞。
Ji-Xin Cheng 教授表示,SRS-FISH 能够以每细胞 10 到 100 毫秒的速度对细胞特性和代谢进行相关成像。相比之下,传统的自发拉曼 FISH记录单个细胞的拉曼光谱大约需要 20 秒才能完成。
“目前为止,微生物学家只能在单细胞水平上研究微生物的功能,SRS-FISH成像速度的大幅提升意味着现在也可以使用较少的微生物组进行单细胞同位素测量”,维也纳大学研究团队的主要负责人Michael Wagner说道。
为了证明 SRS-FISH 将系统发育特性(基因型)与代谢活性(表型)联系起来的能力,研究人员在重水(即氧化氘)中培养样品,使氘能够融入代谢活跃的细胞中。重水中的氘掺入可以与基于拉曼的方法(如 SRS-FISH)相结合,以跟踪单细胞水平对各种化合物的代谢活动。SRS-FISH 能够快速、灵敏地测定单个细胞的氘含量,同时揭示它们的系统发育特性。
研究人员将该方法应用于复杂的微生物群落,并局部追踪了人类肠道中两组微生物的代谢反应,即拟杆菌和梭状芽胞杆菌。多个样本的代谢和特性分析表明,梭状芽胞杆菌对黏膜糖降解的贡献可能比原先认为的要大。
实验结果表明,SRS-FISH 具有识别微生物群中特定微生物代谢的潜力。“将 SRS-FISH 应用于肠道微生物组证明了我们的方法适用于将身份识别与复杂微生物群落内的代谢联系起来。同时,它还揭示了一些有趣的发现,这些发现与人类肠道中黏膜糖的觅食有关”Cheng 说。
Wagner 认为深入了解人体微生物群的功能对更好地理解其在人类健康中的作用和开发更有针对性的益生菌至关重要。
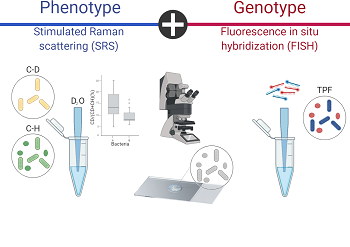
图2 SRS 和 FISH 组合图。SRS 提供的高速、高溶液化学图谱提供了有关代谢的信息,并且该方法提供了定量分析和荧光技术的兼容性。 FISH 提供原位功能,是一种非破坏性方法。
研究人员认为,对 FISH 靶向细胞进行 SRS 选择性扫描可以进一步提高 SRS-FISH 的通量,尤其是在目标分类群数量极少的情况下。具有升级波长调谐的激光设备也可能会增加吞吐量。通过实施可见 SRS,可以提高 SRS-FISH 的灵敏度和分辨率。
SRS-FISH可以应用于范围广泛的环境样品(例如,海洋沉积物、土壤),包括存在一些身荧光背景的样品,因为SRS比自发拉曼光谱对样品自身荧光更具弹性。同时,SRS-FISH不仅限于微生物样本,还适用于真核生物。
Cheng 表示,“在环境中对单个微生物进行成像的传统方法(例如单细胞同位素探测)一次只能分析几个细胞,如果使用SRS-FISH ,能够以快速且精确扫描多个样本,抓住现有低通量方法可能遗漏的细节,更全面地了解微生物在自然环境中的功能。”
该研究发表在 PNAS (www.doi.org/10.1073/pnas.2203519119)。
 客服热线:
客服热线:














